Que recherchez-vous ?
Un contenu sur ce site
Une personne sur l'annuaire
Une formation sur le catalogue
Un contenu sur ce site
Une personne sur l'annuaire
Une formation sur le catalogue
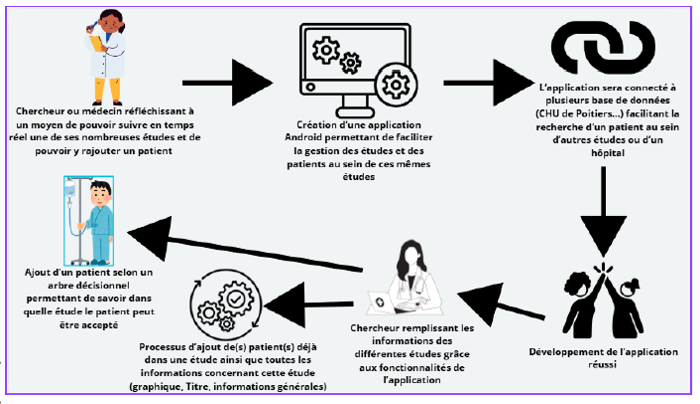
Résumé du projet :
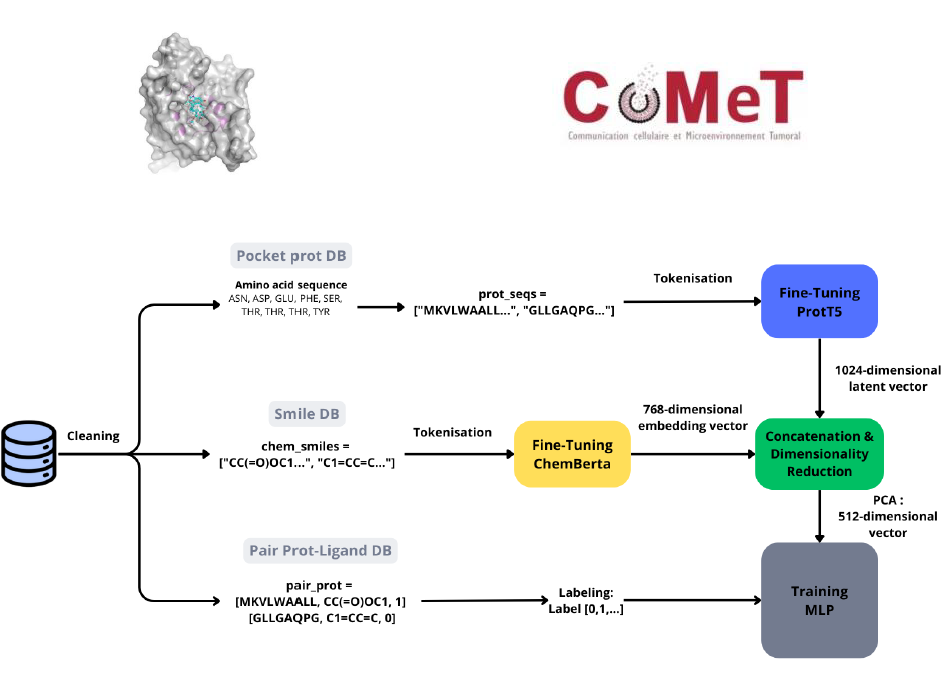
Le projet Kinase IA, développé en partenariat avec le laboratoire COMET, spécialisé en cancérologie, vise à améliorer la présélection « in silico » de molécules candidates pour des tests « in vitro ». Grâce à l’intelligence artificielle et au deep learning, ce projet propose une approche innovante pour identifier des ligands susceptibles d’interagir avec des protéines cibles, en s’appuyant sur des méthodes avancées d’extraction de caractéristiques.
La méthodologie repose sur une architecture combinant plusieurs modèles spécialisés. Les protéines et les ligands sont transformés en représentations numériques optimisées, affinées pour maximiser la pertinence des caractéristiques et améliorer la structuration des familles chimiques. Ces représentations sont ensuite fusionnées et analysées par un réseau de neurones multicouche (MLP) chargé de prédire les interactions potentielles.
Titre du projet: Modélisation cellulaire
Résumé:
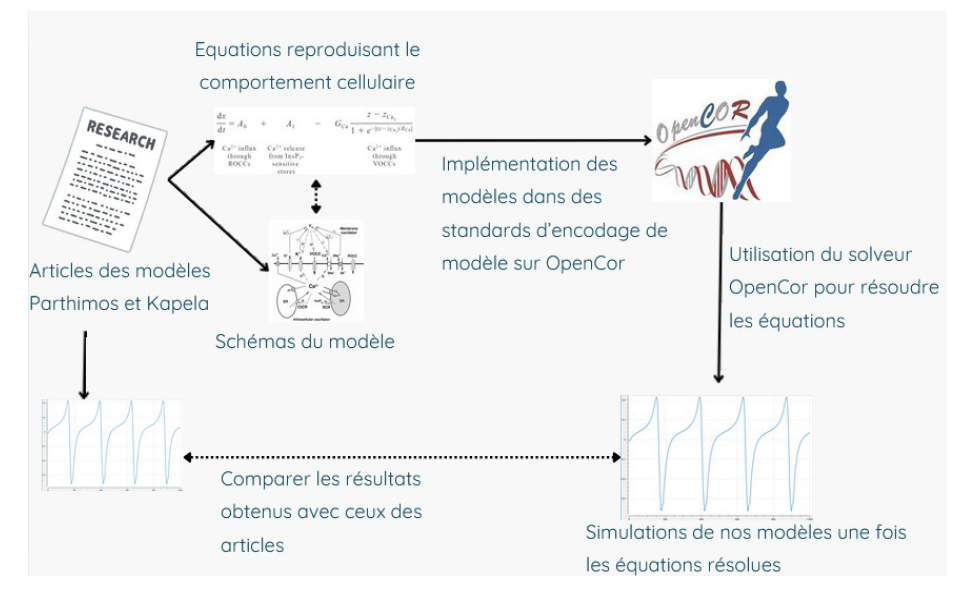
L’objectif du projet était de développer deux modèles numériques des cellules musculaires lisses vasculaires à partir d’articles scientifiques, sur le logiciel OpenCor. Une fois les modèles implémentés, ils seront utilisés dans les TP d’université afin de remplacer les animaux lors des tests de l’effet de certaines substances pharmacologiques sur des types cellulaires. Nous avons commencé par développer le modèle issu de l’article Parthimos, qui permettait de s’entraîner avant de développer le modèle issu de l’article Kapela, qui est bien plus complexe et détaillé (c’est celui-ci qui sera utilisé dans les TP). Nous avons terminé et validé les deux modèles. La validation des modèles s’est faite d’une part en comparant les courbes obtenues sur OpenCor avec celles présentes dans l’article. D’autre part, OpenCor contient un outil qui permet de vérifier la cohérence du code, des équations, des unités… Une fois le travail terminé, nous avons rédigé des documentations notamment sur la façon de développer un modèle fonctionnel et sur les détails pour comprendre l’implémentation de nos modèles. Nous avons publié nos deux modèles sur «https://models.physiomeproject.org» afin de partager notre travail avec la communauté scientifique. Enfin, nous avons pu aller à Tours voir nos commanditaires et voir l’utilisation du modèle Kapela dans un contexte de TP.